理論創薬研究所の金子信人です。
今回は物質名から化合物情報を取得するMoleculeResolverを紹介します。
参考論文
How to crack a SMILES: automatic crosschecked chemical structure resolution across multiple services using MoleculeResolver
(Simon Müller, Journal of Cheminformatics 2025, 17, 117.)
https://doi.org/10.1186/s13321-025-01064-7
SMILES(simplified molecular input line entry system)は分子構造を英数字記号の文字列で表記したもので、少ないデータ量で分子を扱うことができるためデータサイエンスでは広く用いられています。一方で物質名からSMILES情報を取得するためにはCAS※1やPubChem※2などの化合物データベースを検索するのが一般的ですが、データベースによって情報の有無の差があったり、複数の名称がある物質では書き方によってはヒットしないなどの問題がありました。今回紹介するMoleculeResolverは複数のデータベースからクロスチェックすることにより高精度で化合物情報を取得することを可能にしたものになります。
動作環境
Intel Core i7-1265U
Windows 11 Pro
conda 25.7.0
pip 25.2
CheméoのAPI取得
データベースの一つであるCheméoからはAPIを経由して情報を取得していますので、先にAPIを取得します。https://www.chemeo.com/ にアクセスし、右上のメニューからRegisterをクリックし登録します。
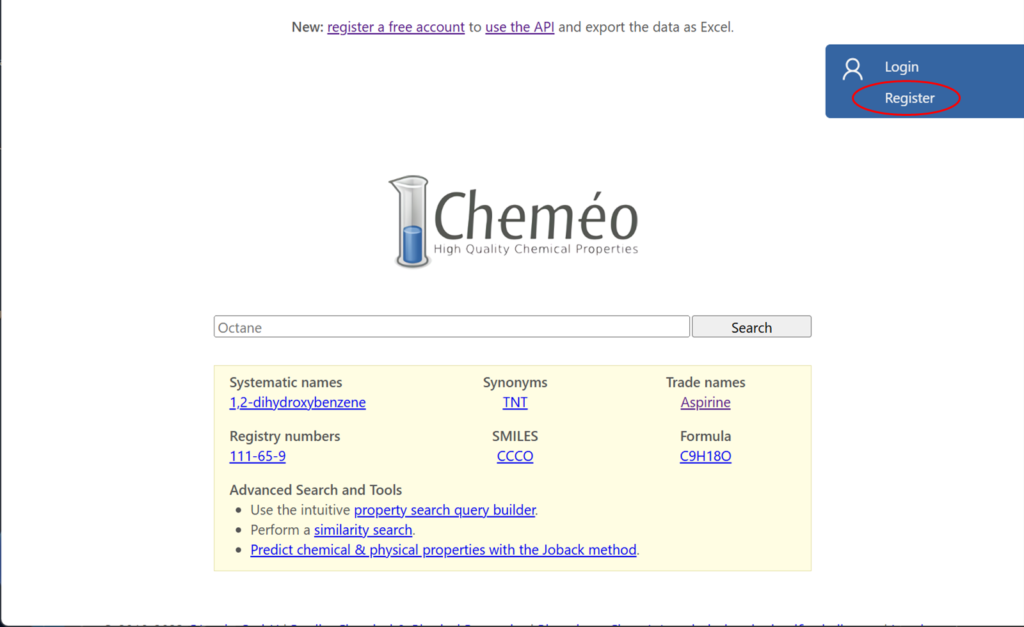
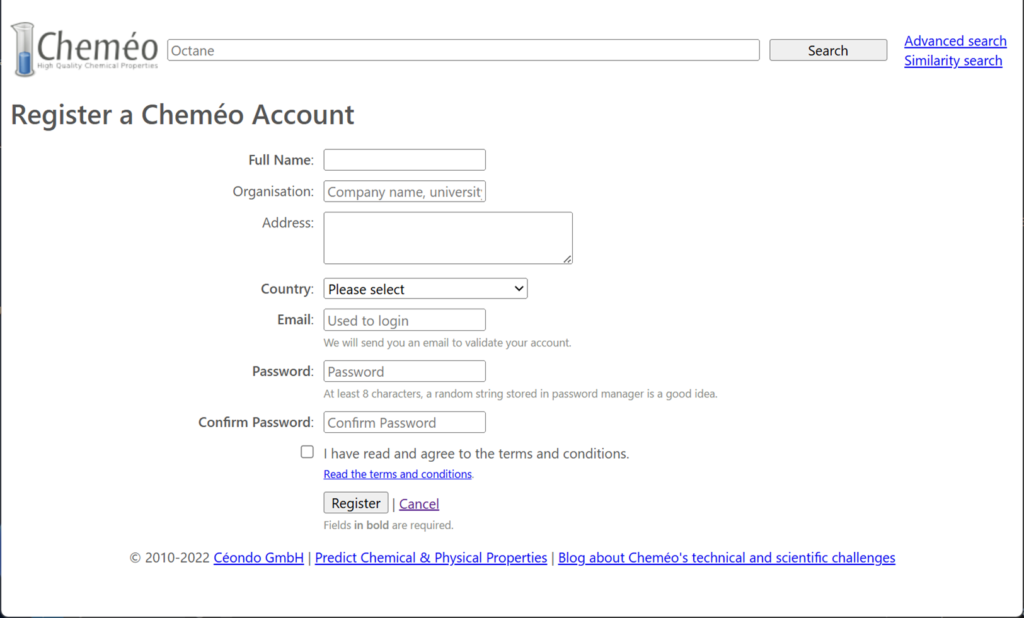
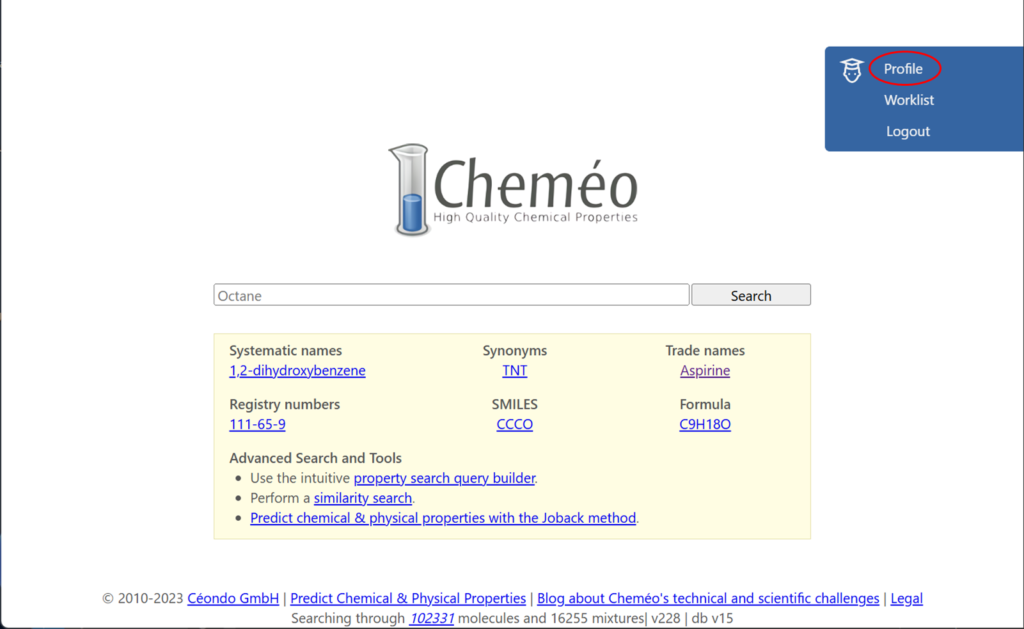
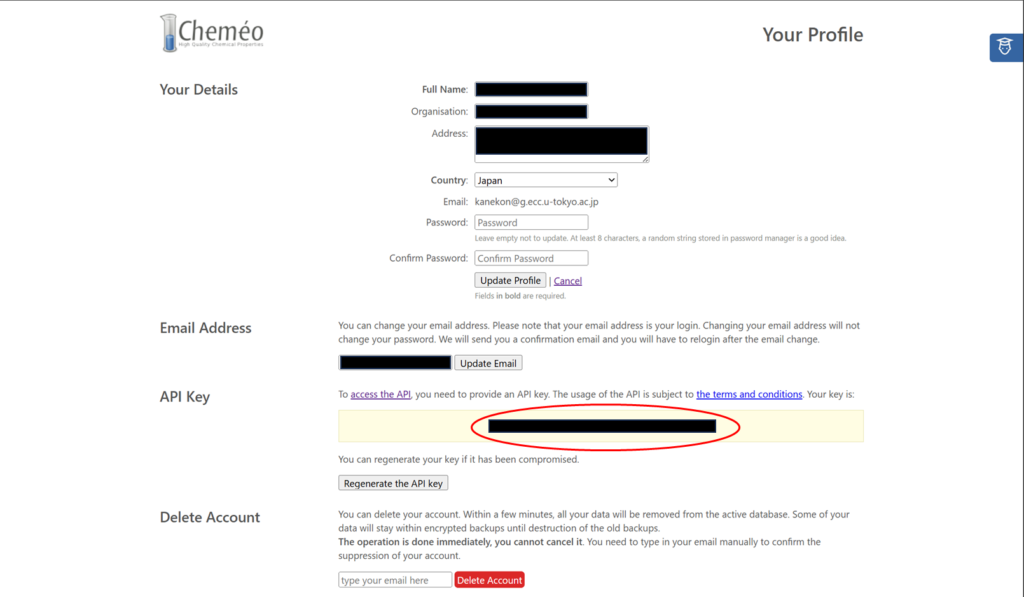
名前・国・メールアドレス・パスワードを入力して登録すると、記入したメールアドレスにメールが届きますので、メールに記載されたリンクからログインします。ログイン後右上のメニューからProfileを選択すると中央にAPI Keyが表示されますのでコピーしておきます。
MoleculeResolverのインストール
Pythonの仮想環境を作成し、MoleculeResolverをインストールします。
$ conda create -n MoleculeResolver python=3.12 -y
$ conda activate MoleculeResolver
$ pip install molecule-resolvernumpy, pillow, rdkitなどの依存パッケージが自動でインストールされました。
MoleculeResolverの実行
Pythonの対話モードでMoleculeResolverを実行してみましょう。
YOUR_API_KEYの部分に先ほど取得したAPI Keyを入力します。
>>> from rdkit import Chem
>>> from moleculeresolver import MoleculeResolver
>>> with MoleculeResolver(available_service_API_keys={"chemeo": "YOUR_API_KEY"}) as mr:
... molecule_name = mr.find_single_molecule(["aspirin"], ["name"])
...
>>> print(molecule_name)
Molecule(SMILES='CC(=O)Oc1ccccc1C(=O)O', synonyms=['aspirin', '2-acetyloxybenzoic acid', '2-Acetoxybenzoic acid', '2-(Acetyloxy)benzoic acid', '2-Carboxyphenyl acetate'], CAS=[], additional_information='cir', mode='name', service='cir', number_of_crosschecks=1, identifier='aspirin', found_molecules=[])分子名検索で入力したaspirinに対し、SMILESと別称・付随情報が出力されます。
複数の分子を同時に検索することも可能で、GitHubで提示されているPythonスクリプトを実行してみましょう。
import json
from moleculeresolver import MoleculeResolver
molecule_names = ["aspirin", "propanol", "ibuprofen", "non-exixtent-name"]
not_found_molecules = []
molecules_dicts = {}
with MoleculeResolver(available_service_API_keys={"chemeo": "YOUR_API_KEY"}) as mr:
molecules = mr.find_multiple_molecules_parallelized(molecule_names, [["name"]] * len(molecule_names))
for name, molecule in zip(molecule_names, molecules):
if molecule:
molecules_dicts[name] = molecule.to_dict(found_molecules='remove')
else:
not_found_molecules.append(name)
with open("molecules.json", "w") as json_file:
json.dump(molecules_dicts, json_file, indent=4)
print(f"Molecules not found: {not_found_molecules}")実行するとコンソールには以下のように表示され、’non-exixtent-name’ という名前の分子は存在しないというように分別されます。
Getting data from services
100%|█████████████████| 4/4 [00:10<00:00, 2.59s/it]
Molecules not found: ['non-exixtent-name']実行結果はmolecules.jsonに保存され、検索した分子のSMILES・別称・CAS登録番号といくつのデータベースでクロスチェックされたか等が出力されます。
{
"aspirin": {
"SMILES": "CC(=O)Oc1ccccc1C(=O)O",
"synonyms": [
"aspirin",
"2-(acetyloxy)-Benzoic acid",
"2-acetyloxybenzoic acid",
"acetylsalicylic acid",
"2-Acetoxybenzoic acid"
],
"CAS": [
"50-78-2"
],
"additional_information": "cir; nist id: C50782; pubchem id: 2244; srs id: 1198",
"mode": "name; name; name; name",
"service": "cir; nist; pubchem; srs",
"number_of_crosschecks": 4,
"identifier": "aspirin"
},
"propanol": {
"SMILES": "CCCO",
"synonyms": [
"propanol",
"1-Propanol",
"Alcohol, propyl",
"Propan-1-ol",
"Propyl alcohol"
],
"CAS": [
"71-23-8"
],
"additional_information": "chemeo id: 15-582-9; cir; nist id: C71238; ; pubchem id: 1031; srs id: 17121898",
"mode": "name; name; name; name; name; name",
"service": "chemeo; cir; nist; opsin; pubchem; srs",
"number_of_crosschecks": 6,
"identifier": "propanol"
},
"ibuprofen": {
"SMILES": "CC(C)Cc1ccc(C(C)C(=O)O)cc1",
"synonyms": [
"ibuprofen",
"2-[4-(2-methylpropyl)phenyl]propanoic acid",
"2-(4-Isobutylphenyl)propanoic acid",
"(.+/-.)-2-(p-Isobutylphenyl)propionic acid",
"(.+/-.)-p-Isobutylhydratropic acid"
],
"CAS": [
"15687-27-1"
],
"additional_information": "chemeo id: 20-897-4; cir; nist id: T999947192; ; pubchem id: 3672; srs id: 200931",
"mode": "name; name; name; name; name; name",
"service": "chemeo; cir; nist; opsin; pubchem; srs",
"number_of_crosschecks": 6,
"identifier": "ibuprofen"
}
}より使いやすいようにCSVファイルを読み込んで、一列目にある化合物名を検索して二列目にSMILESを書き込むコードを作成してみます。同様にYOUR_API_KEYの部分に取得したAPI Keyを入力してください。
import csv
from moleculeresolver import MoleculeResolver
# Input and output file names
input_filename = 'molecule.csv'
output_filename = 'molecule_resolved.csv'
# Read molecule names from the CSV
molecule_names = []
try:
with open(input_filename, 'r', newline='', encoding='utf-8') as infile:
reader = csv.reader(infile)
header = next(reader)
for row in reader:
if row:
molecule_names.append(row[0])
except FileNotFoundError:
print(f"Error: Input file '{input_filename}' not found.")
not_found_molecules = []
output_data = [['name', 'SMILES']]
with MoleculeResolver(available_service_API_keys={"chemeo": "YOUR_API_KEY"}) as mr:
molecules = mr.find_multiple_molecules_parallelized(
molecule_names,
[["name"]] * len(molecule_names)
)
for name, molecule in zip(molecule_names, molecules):
if molecule and molecule.SMILES:
output_data.append([name, molecule.SMILES])
else:
output_data.append([name, ''])
not_found_molecules.append(name)
with open(output_filename, 'w', newline='', encoding='utf-8') as outfile:
writer = csv.writer(outfile)
writer.writerows(output_data)
print(f"Processing complete. Results are saved in '{output_filename}'.")
if not_found_molecules:
print(f"Molecules not found: {not_found_molecules}")実行するとmolecule.csvを読み込んでSMILESの書き込まれたmolecule_resolved.csvを出力します。
| name | SMILES |
| imatinib | Cc1ccc(NC(=O)c2ccc(CN3CCN(C)CC3)cc2)cc1Nc1nccc(-c2cccnc2)n1 |
| gefitinib | COc1cc2ncnc(Nc3ccc(F)c(Cl)c3)c2cc1OCCCN1CCOCC1 |
| ibrutinib | C=CC(=O)N1CCCC@@Hc3c(N)ncnc32)C1 |
| tetrodotoxin | N=C1NC(O)C2C3OC4(O)OC(C(O)C2(N1)C4O)C3(O)CO |
| paclitaxel | CC(=O)OC1C(=O)C2(C)C(O)CC3OCC3(OC(C)=O)C2C(OC(=O)c2ccccc2)C2(O)CC(OC(=O)C(O)C(N=C(O)c3ccccc3)c3ccccc3)C(C)=C1C2(C)C |
| manzamine a | O[C@]12C=C(c3nccc4c3[nH]c3ccccc34)[C@@H]3CCN(CCCC/C=C\CC1)C[C@@]31CC3/C=C\CCCCN3[C@H]12.[Cl-].[H+] |
医薬品化合物や天然物などもきちんと検索できますので、精度が高いことがわかります。
おわりに
構造情報とSMILESは対応関係があるので変換ソフトウェアを使用することで簡単にアクセスすることができる一方で、化合物名は複数の名称が混在しているためデータベースからの検索が困難でした。今回紹介したMoleculeResolverはクロスチェック機能により高精度でSMILES情報を取得できます。化合物名のみのデータベースからバーチャルスクリーニング用にSMILES情報を取得したいときに有用になるかと思います。
※1 CAS(Chemical Abstracts Service)アメリカ化学会の運営する化合物データベース
https://www.cas.org/ja
※2 PubChem アメリカ国立衛生研究所の運営する化合物データベース
https://pubchem.ncbi.nlm.nih.gov/
前回記事
Chempropを活用した機械学習による化学特性予測
https://www.insilico.jp/blog/2025/09/11/chemprop/
Category: AI創薬関連