理論創薬研究所の金子信人です。
今回は分子類似性評価ソフトウェアのGESimの紹介をしていきます。
参考文献
GESim: ultrafast graph-based molecular similarity calculation via von Neumann graph entropy
(Hiroaki Shiokawa, Shoichi Ishida, Kei Terayama, Journal of Cheminformatics, 2025, 17, 57)
https://jcheminf.biomedcentral.com/articles/10.1186/s13321-025-01003-6
動作環境
Intel Core i7-1265U
Windows 11 Pro
conda 25.3.1
分子類似性とは対象となる分子同士が、構造的・機能的にどの程度似通っているかという指標になります。これは「類似した化合物は類似した性質を持つ (similar property principle)」※1という概念に基づいており、創薬研究においては既存薬との類似性を評価することで同等の薬効を示す化合物の探索などに用いられています。この類似性の評価には様々な指標が用いられており、もっとも代表的なTanimoto係数では分子構造をフィンガープリントにし、その一致度合いを評価するものになります。今回紹介するGESimではvon Neumann graph entropyを用いて分子の類似性を評価します。
GESimのインストール
Pythonの仮想環境を作成し、GESimをインストールします。
$ conda create -n GESim python=3.11 -y
$ conda activate GESim
$ pip install --upgrade git+https://github.com/LazyShion/GESim.git必要なパッケージが自動でインストールされ、本環境ではPillow-11.2.1 gesim-0.0.1 numpy-1.26.4 pybind11-2.11.2 rdkit-2023.9.6がインストールされました。
GESimの実行
チュートリアルにしたがって使ってみましょう。
Pythonの対話モードでGESimをインポートし、化合物情報をSMILES表記で入力します。
$ Python
>>> from rdkit import Chem
>>> from gesim import gesim
>>> mol1 = Chem.MolFromSmiles('Cc1nn(C2CCN(Cc3cccc(C#N)c3)CC2)cc1-c1ccccc1')
>>> mol2 = Chem.MolFromSmiles('Cc1nn(C2CCN(Cc3cccc(Cl)c3)CC2)cc1-c1ccccc1')
>>> print(gesim.graph_entropy_similarity(mol1, mol2))
0.9580227325517037mol1に対し、mol2は0.958の類似度を有していると評価されました。スコアは0-1に正規化され、完全に一致している場合は1になります。
複数分子を一度に評価したい場合はbatchを用いて、
>>> mols = [mol1, mol2, Chem.MolFromSmiles('c1ccc(CN2CCC(n3nccc3-c3ccccc3)CC2)cc1')]
>>> print(gesim.graph_entropy_similarity_batch(mol1, mols))
[1.0, 0.9580227325517037, 0.7766178810633495]とするとmolsに含まれるすべての分子に対して、mol1との類似性が評価されました。
一致している部分構造を描画してみましょう。
Jupyter Notebookで実行している場合は以下のコマンドで、
>>> from rdkit.Chem import Draw
>>> mol1_matched, mol2_matched = gesim.get_matched_mapping_numbers(mol1, mol2)
>>> mol1_hal = [i for i, mv in enumerate(mol1_matched) if mv == 1]
>>> mol2_hal = [i for i, mv in enumerate(mol2_matched) if mv == 1]
>>> Draw.MolsToGridImage([mol1, mol2], highlightAtomLists=[mol1_hal, mol2_hal], molsPerRow=2, useSVG=True, subImgSize=(250, 200))Windows PowerShell等で実行している場合は最終行を、
>>> img = Draw.MolsToGridImage([mol1, mol2], highlightAtomLists=[mol1_hal, mol2_hal], molsPerRow=2, useSVG=False, subImgSize=(250, 200))
>>> img.show()として実行します。
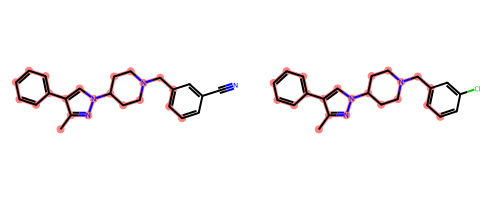
結果はこのように一致している原子がハイライトされて表示されます。
GESimではどういったパラメータが重視されるのか、いくつかの分子を比較してみましょう。
まずはベンゼンの置換体を評価してみます。
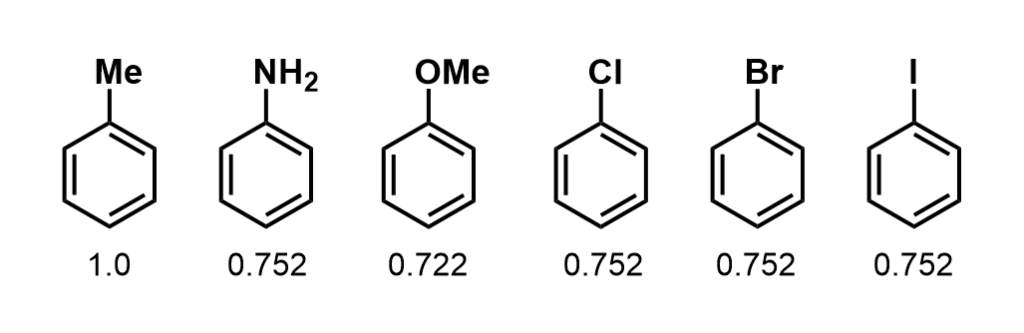
トルエンに対しアニリンでは0.752と評価されました。アニソールでは重原子が1つ多いためかやや低くなりました。一方ハロゲン置換体ではすべて同じスコアで評価されました。このことから電子密度や原子サイズなどは類似性の評価に含まれていないことが推察されます。
次にBioisostere(生物学的等価体)について評価してみます。
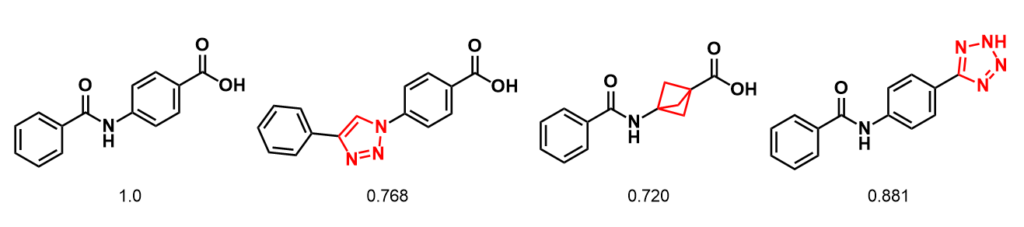
代表的なBioisostereとしてアミド結合をトリアゾールに、ベンゼンをビシクロペンタンに、カルボン酸をテトラゾールに変換したものを評価しました。いずれの場合も等価体による類似性評価の加点は低いようです。スコアの違いは前者2つが構造中心のあるのに対し、後者は構造末端にあるためと推察されます。
おわりに
今回紹介したGESimは非常に高速で、ラップトップパソコンでも瞬時に結果が出力されます。そのため大規模データベースからのスクリーニングにおいても有用だと思われます。一方で化合物の電子密度や嵩高さは類似性に評価されないことから、そういったパラメータを重視する必要がある場合は別のソフトウェアを選択する必要があります。
※1 Johnson MA, Maggiora GM, (1990) Concepts and applications of molecular similarity. Wiley, New York.
前回記事
AiZynthFinderによる逆合成解析
https://www.insilico.jp/blog/2025/06/05/aizynthfinder_retrosynthesis/
次回記事
Direct MaxFlux法による遷移状態予測
https://www.insilico.jp/blog/2025/07/10/direct-maxflux/
Category: AI創薬関連