理論創薬研究所の金子信人です。
今回はオープンソースの逆合成解析ソフトウェアのAiZynthFinderを紹介します。逆合成解析とはある化合物を合成するときに、どんな反応を用いてどんな原料を使えばいいのかという考え方で、有機合成化学において非常に重要な概念になります。エレガントな逆合成解析ができれば、目的とする化合物を簡単に安価に合成できるわけですから、創薬や材料化学にも密接にかかわってきます。
参考論文
AiZynthFinder: a fast, robust and flexible open-source software for retrosynthetic planning
(Samuel Genheden; Amol Thakkar; Veronika Chadimová; Jean-Louis Reymond; Ola Engkvist; Esben Bjerrum. Journal of Cheminformatics, 2020, 12, 70.)
https://jcheminf.biomedcentral.com/articles/10.1186/s13321-020-00472-1
AiZynthFinder 4.0: developments based on learnings from 3 years of industrial application
(Lakshidaa Saigiridharan; Alan Kai Hassen; Helen Lai; Paula Torren-Peraire; Ola Engkvist; Samuel Genheden. Journal of Cheminformatics, 2024, 16, 57.)
https://jcheminf.biomedcentral.com/articles/10.1186/s13321-024-00860-x
動作環境
Intel Core i7-1265U
Windows 11 Pro
conda 25.3.1
逆合成解析は有機合成の大家であるElias Coreyが提唱した概念であり、その功績から1990年にノーベル化学賞を受賞しています。コンピュータによる逆合成解析の試みは当時から行われており、Coreyらは1971年にLHASA(Logic and Heuristics Applied to Synthetic Analysis)というプログラムを開発しています。今回紹介するAiZynthFinderはアストラゼネカ社のグループが開発した逆合成解析ソフトウェアでモンテカルロ木探索とニューラルネットワークを用いたアルゴリズムによる解析を行います。
AiZynthFinderのインストール
Pythonの仮想環境を作成し、AiZynthFinderをインストールします。
一部のパッケージはC++で書かれているので、Windowsの場合はVisual Studio(https://visualstudio.microsoft.com/ja/downloads/)のインストールが必要です。
またpipのバージョンが最新の場合インストールにエラーがでますのでダウングレードしています。
$ conda create "python>=3.9,<3.11" -n aizynth-env
$ conda activate aizynth-env
$ python -m pip install --upgrade pip==23.3
$ python -m pip install aizynthfinder[all]インストールが完了したら学習済みモデルとストックコレクションをダウンロードします。
$ download_public_data .config.ymlが作成されればダウンロードは完了です。
AiZynthFinderの実行
AiZynthFinderはCUIでの実行と、Jupyter Notebook上でのGUIでの実行が提供されています。今回はCUIでの実行例を紹介します。
目的化合物のSMILESをsmiles.txtに作成します。例として消炎鎮痛剤のセレコキシブ(O=S(=O)(c3ccc(n1nc(cc1c2ccc(cc2)C)C(F)(F)F)cc3)N)を逆合成解析してみましょう。
$ aizynthcli --config config.yml --smiles smiles.txt
Loading template-based expansion policy model from C:\Users\user\uspto_model.onnx to uspto
Loading templates from C:\Users\user\uspto_templates.csv.gz to uspto
Loading template-based expansion policy model from C:\Users\user\uspto_ringbreaker_model.onnx to ringbreaker
Loading templates from C:\Users\user\uspto_ringbreaker_templates.csv.gz to ringbreaker
Loading filter policy model from C:\Users\user\uspto_filter_model.onnx to uspto
Loading stock from InMemoryInchiKeyQuery to zinc
Selected as stock: zinc
Compounds in stock: 17422831
Selected as expansion policy: uspto
Selected as filter policy: uspto
Done with O=S(=O)(c3ccc(n1nc(cc1c2ccc(cc2)C)C(F)(F)F)cc3)N in 71.9 s and is solved
Output saved to output.json.gz解析結果はoutput.json.gzに出力されます。出力結果のビジュアライズは公式ドキュメントに記載されているPythonスクリプトを保存し、実行します。
import pandas as pd
from aizynthfinder.reactiontree import ReactionTree
data = pd.read_json("output.json.gz", orient="table")
all_trees = data.trees.values # This contains a list of all the trees for all the compounds
trees_for_first_target = all_trees[0]
for itree, tree in enumerate(trees_for_first_target):
imagefile = f"route{itree:03d}.png"
ReactionTree.from_dict(tree).to_image().save(imagefile)実行すると解析結果がpngファイルで出力されます。今回は5つの結果が出力されました。
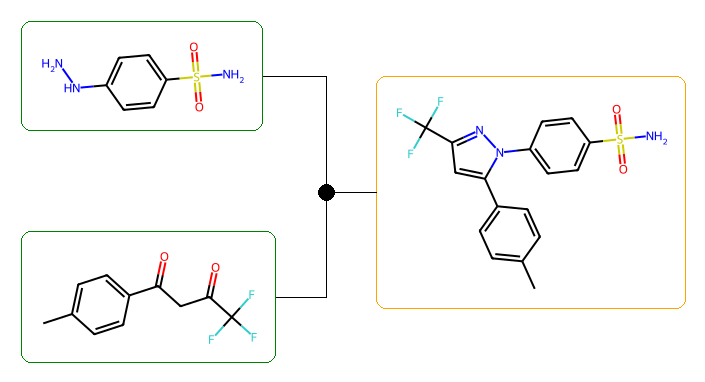
1つ目の結果では実際の合成経路であるβ-ジケトンとヒドラジンの脱水縮合反応が示されています。
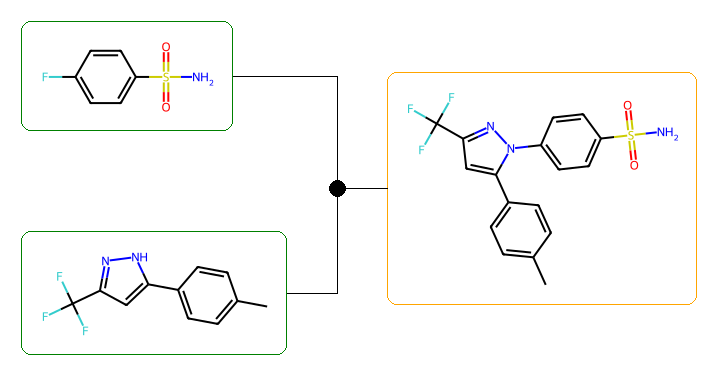
また、4つ目の結果ではピラゾールとフルオロベンゼンスルホンアミドとのSNAr反応が提案されました。
このようにいくつかの合成経路が考えられる場合は複数の結果が出力されます。一方で各反応の反応名および反応試薬等は明示されませんので、基本的な有機合成の知識と文献調査は必要となってきます。
しかしながら複雑な化合物の逆合成解析ではnot solvedとなってしまい適用範囲には課題が残ります。また解析は学習元のデータベースに依存するため、医薬品開発で広く用いられるクロスカップリング反応や縮合反応に強い一方で、環化反応や転位反応はあまり提示されない傾向が見受けられます。
おわりに
AiZynthFinderはX(旧Twitter)のBotである逆合成ちゃん(@retrosynthchan)※1として運用もされており、逆合成したい化合物をSMILES形式でリプライすると逆合成解析を行ってくれます。精度としてはまだまだ発展途上ですが、SciFinderやReaxysなどの商用の科学情報プラットフォームが利用できない場合や、有機合成化学に明るくない場合に手間やコストを見積もるなどの利用には力を発揮しそうです。AI技術の発展が目覚ましい昨今、さらなる改良が期待される分野ですので注視していきたいですね。
※1 Twitter integration of chemistry software tools (Naruki Yoshikawa, Ryuichi Kubo, Kazuki Z. Yamamoto, Journal of Cheminformatics, 2021, 13, 46.)
https://jcheminf.biomedcentral.com/articles/10.1186/s13321-021-00527-x
逆合成ちゃんを支える技術 (https://zenn.dev/retrosynthchan/articles/9283f3e8dd8dac)
前回記事
GNINAを用いたドッキングシミュレーション
https://www.insilico.jp/blog/2025/05/19/gnina_docking/
次回記事
GESimによる分子類似性評価
https://www.insilico.jp/blog/2025/06/19/gesim_similarity/
Category: AI創薬関連