理論創薬研究所の金子信人です。
今回はオープンソースのドッキングシミュレーションソフトウェアを用いて、低分子化合物と標的タンパク質のドッキングを行っていきます。
参考論文
GNINA 1.0: molecular docking with deep learning
(Andrew T. McNutt, Paul Francoeur, Rishal Aggarwal, Tomohide Masuda, Rocco Meli, Matthew Ragoza, Jocelyn Sunseri, David Ryan Koes, Journal of Cheminformatics 2021, 13, 43)
https://jcheminf.biomedcentral.com/articles/10.1186/s13321-021-00522-2
GNINA 1.3: the next increment in molecular docking with deep learning
(Andrew T. McNutt, Yanjing Li, Rocco Meli, Rishal Aggarwal, David Ryan Koes, Journal of Cheminformatics, 2025, 17, 28.)
https://jcheminf.biomedcentral.com/articles/10.1186/s13321-025-00973-x
ドッキングシミュレーションソフトウェアは商用・非商用問わず様々な種類のものが開発されていますが、今回紹介するGNINA(Generative Neural Network for Interactive Applications)は、既存のドッキングソフトウェアであるAutodock VinaやSminaにディープラーニングを統合し、3D-畳み込みニューラルネットワーク(3D-CNN)を用いて、より柔軟で高精度なスコアリングを可能にしたものになります。
動作環境
Intel Core i9-10900K
Nvidia GeForce RTX 3070
Windows 11 home
WSL 2.4.13.0
Ubuntu 24.04
conda 25.3.1
ローカル環境で利用する場合はCUDA>12.0が要求されていますので、Maxwell世代以降のGPU(GTX 750以降)が必要となります。GPU環境がない場合はGoogle Colabで実装されていますので、後ほど使用法を解説します。
GNINAのインストール
GNINAはLinux環境での動作がサポートされています。今回はWindowsでのLinux仮想環境へのインストールを紹介します。まずは仮想環境を作成し、CUDAと関連ソフトウェアをインストールします。
WSLの場合はhttps://docs.nvidia.com/cuda/wsl-user-guide/contents.htmlのガイドにしたがってインストールします。
Linuxの場合はhttps://developer.nvidia.com/cuda-downloadsからdeb (network) を選択してインストール用コマンドを取得します。
$ conda create -n GNINA python=3.12
$ conda activate GNINA
$ sudo apt-key del 7fa2af80
$ wget https://developer.download.nvidia.com/compute/cuda/repos/wsl-ubuntu/x86_64/cuda-wsl-ubuntu.pin
$ sudo mv cuda-wsl-ubuntu.pin /etc/apt/preferences.d/cuda-repository-pin-600
$ wget https://developer.download.nvidia.com/compute/cuda/12.8.1/local_installers/cuda-repo-wsl-ubuntu-12-8-local_12.8.1-1_amd64.deb
$ sudo dpkg -i cuda-repo-wsl-ubuntu-12-8-local_12.8.1-1_amd64.deb
$ sudo cp /var/cuda-repo-wsl-ubuntu-12-8-local/cuda-*-keyring.gpg /usr/share/keyrings/
$ sudo apt-get update
$ sudo apt-get -y install cuda-toolkit-12-8
$ wget https://developer.download.nvidia.com/compute/cudnn/9.8.0/local_installers/cudnn-local-repo-ubuntu2404-9.8.0_1.0-1_amd64.deb
$ sudo dpkg -i cudnn-local-repo-ubuntu2404-9.8.0_1.0-1_amd64.deb
$ sudo cp /var/cudnn-local-repo-ubuntu2404-9.8.0/cudnn-*-keyring.gpg /usr/share/keyrings/
$ sudo apt-get update
$ sudo apt-get -y install cudnn-cuda-12
$ echo 'export PATH=/usr/local/cuda-12.8/bin${PATH:+:${PATH}}' >> ~/.bashrc
$ echo 'export LD_LIBRARY_PATH=/usr/local/cuda-12.8/lib64:${LD_LIBRARY_PATH:+:${LD_LIBRARY_PATH}}' >> ~/.bashrc
$ source ~/.bashrc続いてOpenBabelと必要なソフトウェアのインストールをしていきます。今回はPython関連ソフトウェアはCondaからインストールしています。
$ conda activate GNINA
$ conda install -c conda-forge numpy=1.26 pytest -y
$ apt-get install build-essential git cmake wget swig libboost-all-dev libeigen3-dev libgoogle-glog-dev libprotobuf-dev protobuf-compiler libhdf5-dev libatlas-base-dev librdkit-dev libjsoncpp-dev
$ git clone https://github.com/openbabel/openbabel.git
$ cd openbabel
$ mkdir build
$ cd build
$ cmake -DWITH_MAEPARSER=OFF -DWITH_COORDGEN=OFF -DPYTHON_BINDINGS=ON -DRUN_SWIG=ON ..
$ make -j8
$ sudo make install
$ source ~/.bashrc最後にGNINAのインストールをしていきます。cmakeがcuDNNのPATHを参照できなかったのでPATHを明示しています。
$ conda activate GNINA
$ cd ~
$ git clone https://github.com/gnina/gnina.git
$ cd gnina
$ mkdir build
$ cd build
$ cmake -DCUDNN_INCLUDE_PATH=/usr/include -DCUDNN_LIBRARY_PATH=/usr/lib/x86_64-linux-gnu/libcudnn.so ..
$ make -j8
$ sudo make installgninaと入力して以下の画面が表示されればインストール完了です。
$ gnina
_
(_)
__ _ _ __ _ _ __ __ _
/ _` | '_ \| | '_ \ / _` |
| (_| | | | | | | | | (_| |
\__, |_| |_|_|_| |_|\__,_|
__/ |
|___/
gnina master:25e64da Built Apr 28 2025.
gnina is based on smina and AutoDock Vina.
Please cite appropriately.ドッキングシミュレーションの実行
チュートリアルに記載されているMAPKの阻害剤のドッキングシミュレーションを実行してみましょう。
PDBから構造情報を取得し、レセプターとリガンドをそれぞれ保存し、GNINAを使用してドッキングします。コマンドはそれぞれ -r がレセプターの指定、-l がリガンドの指定、–autobox_ligand はドッキング範囲の指定で今回はすでにリガンド分子が入っている領域を指定しています、-o はアウトプットファイルの指定、–seed はシード値の指定で入力しない場合ランダムシードとなります。
$ wget http://files.rcsb.org/download/3ERK.pdb
$ grep ATOM 3ERK.pdb > rec.pdb
$ grep SB4 3ERK.pdb > lig.pdb
$ gnina -r rec.pdb -l lig.pdb --autobox_ligand lig.pdb -o docked.sdf --seed 0
_
(_)
__ _ _ __ _ _ __ __ _
/ _` | '_ \| | '_ \ / _` |
| (_| | | | | | | | | (_| |
\__, |_| |_|_|_| |_|\__,_|
__/ |
|___/
gnina master:25e64da Built Apr 28 2025.
gnina is based on smina and AutoDock Vina.
Please cite appropriately.
Commandline: gnina -r rec.pdb -l lig.pdb --autobox_ligand lig.pdb -o docked.sdf --seed 0
==============================
*** Open Babel Warning in PerceiveBondOrders
Failed to kekulize aromatic bonds in OBMol::PerceiveBondOrders
Using random seed: 0
0% 10 20 30 40 50 60 70 80 90 100%
|----|----|----|----|----|----|----|----|----|----|
***************************************************
mode | affinity | intramol | CNN | CNN
| (kcal/mol) | (kcal/mol) | pose score | affinity
-----+------------+------------+------------+----------
1 -8.50 -0.79 0.9777 7.179
2 -8.51 -0.84 0.9650 7.199
3 -8.30 -0.72 0.4390 6.297
4 -7.68 -0.65 0.3015 5.951
5 -6.12 -0.53 0.2668 6.026
6 -7.59 -0.65 0.2356 5.685
7 -6.92 -0.47 0.2143 5.956
8 -6.36 -0.95 0.2086 5.438
9 -6.90 -0.77 0.1767 4.250計算が実行されると生成されたコンフォメーションのアフィニティーとスコアが表示され、結果はdocked.sdfに出力されます。出力された構造をPyMOLを使って表示してみましょう。
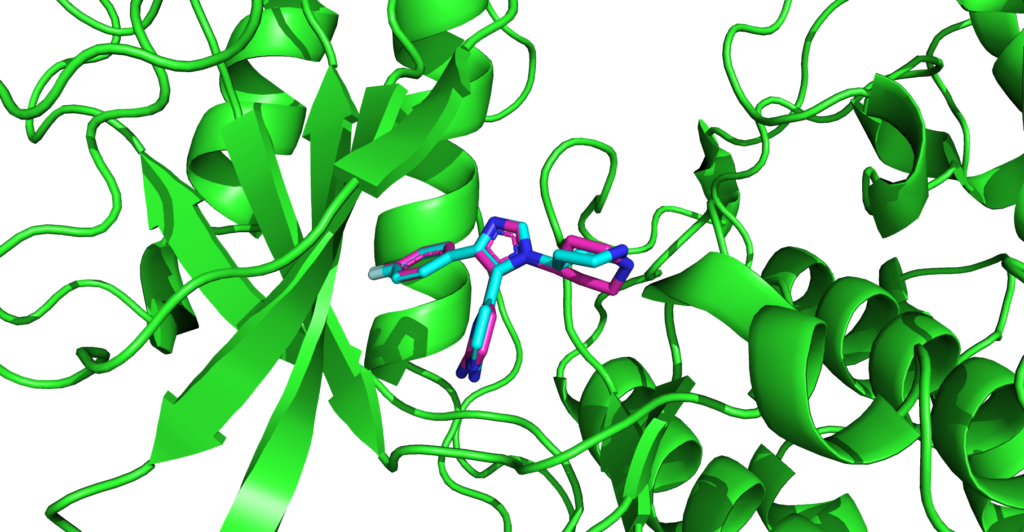
タンパク質構造(PDB:1ERK), ピンク:結晶構造中のリガンド, 青:ドッキングされたリガンド
結晶構造と非常に近い形のドッキング結果が得られました。
コバレントドッキングシミュレーションの実行
GNINA1.3には共有結合性阻害剤のドッキング機能が実装されています。例としてペニシリン結合タンパク質とセフロキシムのコバレントドッキングシミュレーションを実行してみましょう。同様にPDBから構造情報を取得し、レセプターとリガンドをそれぞれ保存し、GNINAを使用してドッキングします。
$ wget http://files.rcsb.org/download/1QMF.pdbダウンロードしたPDBファイルをPyMOLで開き、水分子とリガンド分子を削除したものをレセプターとして、セフロキシムのみをリガンドとしてそれぞれ保存します。コバレントドッキングの場合は共有結合を形成するアミノ酸残基とリガンドの原子をそれぞれ指定する必要があります。–covalent_rec_atom がレセプターのアミノ酸残基の指定でチェイン:残基番号:原子の形で入力し、原子は該当する場所をPyMOLで選択すると表示されます。–covalent_lig_atom_pattern がリガンドの結合原子でSMART形式で指定します。
$ gnina -r rec.pdb -l lig.pdb --autobox_ligand lig.pdb --covalent_rec_atom A:337:OG --covalent_lig_atom_pattern 'CCCSCCCN' -o docked.sdf --seed 0
_
(_)
__ _ _ __ _ _ __ __ _
/ _` | '_ \| | '_ \ / _` |
| (_| | | | | | | | | (_| |
\__, |_| |_|_|_| |_|\__,_|
__/ |
|___/
gnina master:25e64da Built Apr 28 2025.
gnina is based on smina and AutoDock Vina.
Please cite appropriately.
WARNING: CNN scoring not yet calibrated for covalent docking. Recommend running with --cnn_scoring none
Commandline: gnina -r rec.pdb -l lig.pdb --autobox_ligand lig.pdb --covalent_rec_atom A:337:OG --covalent_lig_atom_pattern CCCSCCCN -o docked.sdf --seed 0
==============================
*** Open Babel Warning in PerceiveBondOrders
Failed to kekulize aromatic bonds in OBMol::PerceiveBondOrders
Using random seed: 0
0% 10 20 30 40 50 60 70 80 90 100%
|----|----|----|----|----|----|----|----|----|----|
***************************************************
mode | affinity | intramol | CNN | CNN
| (kcal/mol) | (kcal/mol) | pose score | affinity
-----+------------+------------+------------+----------
1 -8.78 0.00 0.6755 5.042
2 -8.66 0.00 0.6717 4.659
3 -7.69 0.00 0.6614 4.986
4 -8.97 0.00 0.6596 4.783
5 -8.17 0.00 0.6076 5.013
6 -5.13 0.00 0.5839 5.508
7 -6.74 0.00 0.5415 5.280
8 -8.89 0.00 0.5245 4.588
9 -8.78 0.00 0.4824 4.571同様に出力された構造をPyMOLを使って表示してみましょう。
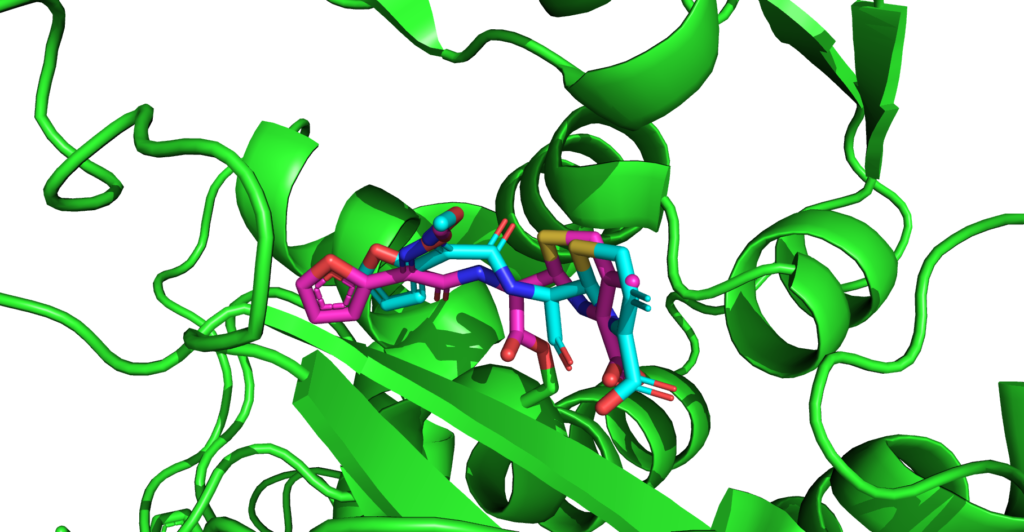
タンパク質構造(PDB:1QMF), ピンク:結晶構造中のリガンド, 青:ドッキングされたリガンド
こちらも結晶構造と非常に近い形のドッキング結果が得られました。
Google Colabでの使用
Colabのページhttps://colab.research.google.com/drive/1QYo5QLUE80N_G28PlpYs6OKGddhhd931?usp=sharingにアクセスします。
右上のその他の接続オプションからランタイムのタイプの変更を選択し、ランタイムのタイプはPython 3をハードウェア アクセラレータはGPUを選択します。ランタイムから全てのセルを実行を選択すると順次実行され、前述のドッキングシミュレーションと同一の結果が出力されます。任意のタンパク質でのドッキングシミュレーションを実行する場合は、PDBから取得するタンパク質を変更するか、用意した構造情報をGoogle driveにアップロードして実行してみてください。
おわりに
これまでドッキングシミュレーションソフトウェアは複雑な入力が必要でしたが、今回紹介したGNINAはシンプルな入力形式に対応しており、計算も非常に高速です。AlphaFoldなどで予測された構造も入力に使用することができますので、創薬研究にお役立てください。
前回記事
BR-SAScoreによる分子の合成難易度予測
https://www.insilico.jp/blog/2025/04/24/br_sascore/
次回記事
AiZynthFinderによる逆合成解析
https://www.insilico.jp/blog/2025/06/05/aizynthfinder_retrosynthesis/
Category: AI創薬関連, Docking