理論創薬研究所の吉森です。
オープンソースのドッキングソフトウェアの多くは、環構造におけるイス形と舟形などの変化を考慮できないため、初期配座がドッキングの結果に与える影響が大きいと思います。今回は、まずは骨格の立体配座を固定した状態で側鎖の立体配座を生成させ、分子の立体配座を生成します。次に、その立体配座を用いてドッキングを行いたいと思います。
骨格の立体配座を固定した初期配座の生成
下図はXIAPと阻害剤の結晶構造(PDB:5OQW)ですが、骨格(炭素:灰色)の立体配座を固定して、側鎖(炭素:黄色)の配座生成を行いたいと思います。
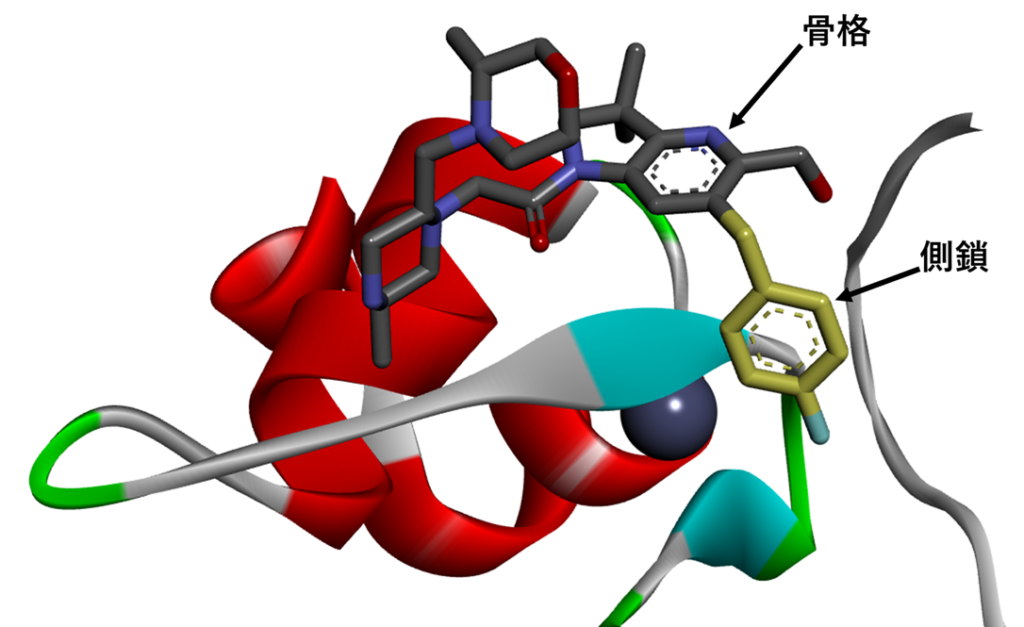
RDKitの”AllChem.ConstrainedEmbed”を利用することで実現できます。
core_embedded.py:
from rdkit.Chem import AllChem
import argparse
parser = argparse.ArgumentParser(description="core_embedded.py")
parser.add_argument("input1", help="mol of core structure")
parser.add_argument("input2", help="smiles of molecules")
parser.add_argument("output", help="sdf of molecules")
def main():
args = parser.parse_args()
core_file = args.input1
smi_file = args.input2
out_file = args.output
core = Chem.MolFromMolFile(core_file)
mols = [Chem.AddHs(mol) for mol in Chem.SmilesMolSupplier(smi_file,delimiter='\t',titleLine=0) if mol is not None]
fw = Chem.SDWriter(out_file)
for mol in mols:
try:
AllChem.ConstrainedEmbed(mol, core)
fw.write(mol)
except:
print(f"{mol.GetProp('_Name')} doesn't have core structure")
fw.close()
if __name__ == '__main__':
main()上記のスクリプトを使って配座生成を行ってみます。
$ python core_embedded.py core.sdf test_cpds.smi test_cpds.sdf
core.sdf:固定したい骨格の立体配座
test_cpds.smi:分子のSMILES(固定したい骨格を部分構造として含んでいること)
test_cpds.sdf:骨格の立体配座が固定された分子の立体配座(出力ファイル)
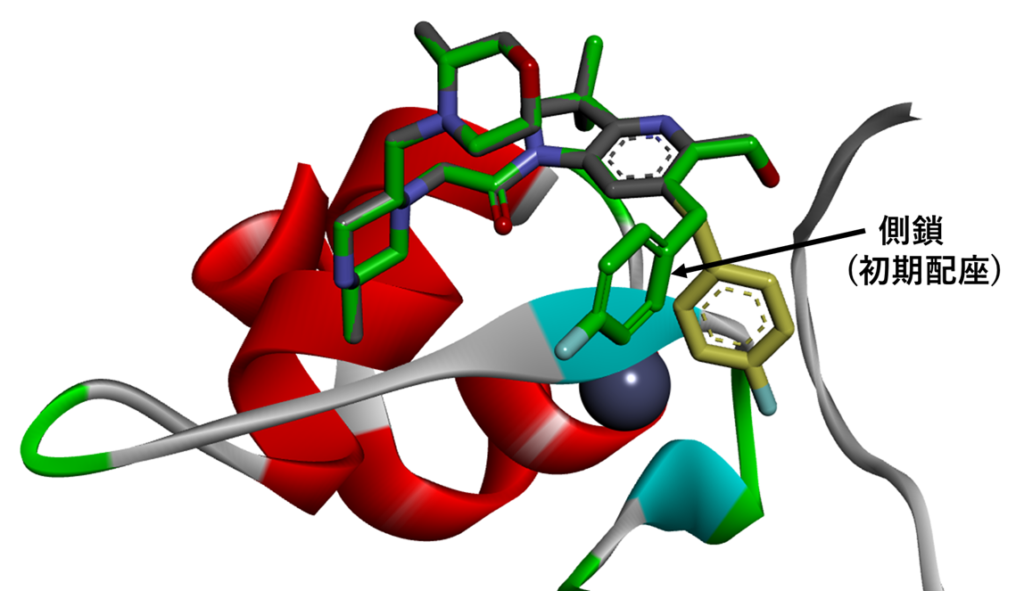
生成された立体配座(炭素:緑)をみると、骨格は結晶構造の立体配座(炭素:灰色)と同じですが、側鎖の立体配座(炭素:黄色)は異なることが分かると思います。
ドッキング
前項で生成した分子の立体配座を用いてGNINAを使ってドキングを行いたいと思います。
$ gnina -r 5OQW_receptor.pdb -l test_cpds.sdf -o docked_test_cpds.sdf –autobox_ligand 5OQW_ligand.sdf –num_modes 1
5OQW_receptor.pdb:XIAPの構造
test_cpds.sdf:前項で生成した分子の立体配座
docked_test_cpds.sdf:ドッキングにより得られた分子の結合配座
5OQW_ligand.sdf:結晶構造に含まれている阻害剤の立体配座
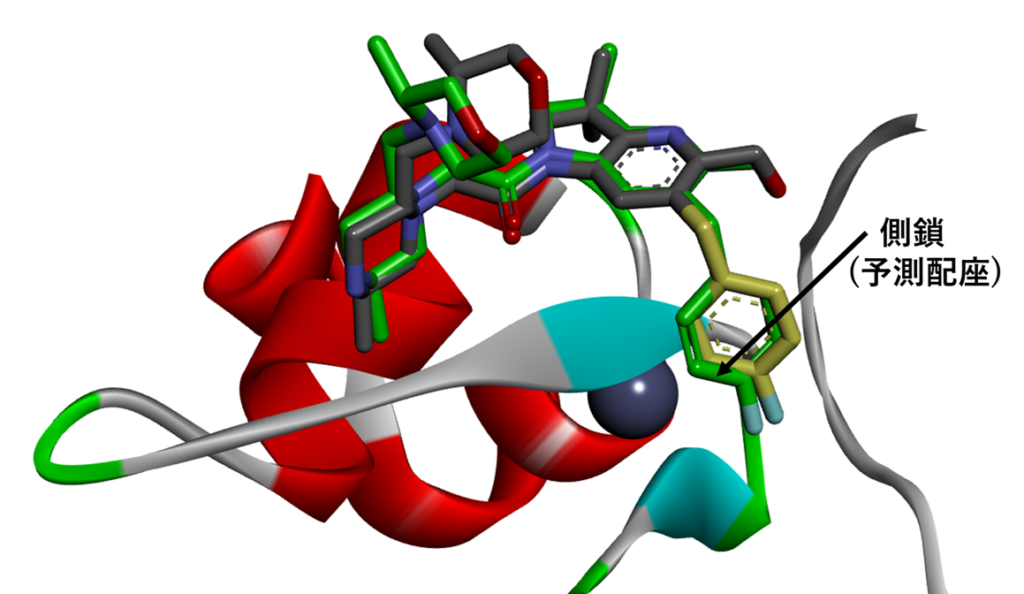
ドッキングによる予測配座(炭素:緑)をみると結晶構造(炭素:灰色 or 黄色)とよく重なっていると思います。今回の手順を用いて、側鎖構造の異なる分子を複数発生させ、ドッキングを行うことで、側鎖構造の最適化などに利用できると思います。
Category: 未分類