理論創薬研究所の吉森です。今回はRDKitを使って簡単な創薬計算用MCPサーバーをWindowsで構築したいと思います。
MCPサーバーの作成
Powershellを管理者権限で起動する。
> powershell -ExecutionPolicy ByPass -c "irm https://astral.sh/uv/install.ps1 | iex"
> uv init rdkit_mcp
> cd rdkit_mcp
> uv venv
> .\.venv\Scripts\activate
> uv add mcp[cli] httpx rdkit
rdkit_mcpフォルダにtpsa_mcp.pyを配置する。
このスクリプトでは、分子のTPSAの計算、並びに各原子のTPSAに対する寄与を計算する2つの関数が実装されている。
tpsa_mcp.py:
from mcp.server.fastmcp import FastMCP
from rdkit import Chem
from rdkit.Chem.Draw import SimilarityMaps,rdMolDraw2D
from rdkit.Chem import rdMolDescriptors,Draw, Descriptors
import os
mcp = FastMCP("atomic_mcp", debug=True)
@mcp.tool()
def atomic_tpsa_fig(smiles):
"""
SMILESからrdkit molオブジェクトに変換をし、TPSAを計算して、画像ファイルを保存する。
原子ごとのTPSAの寄与がわかる。
"""
mol = Chem.MolFromSmiles(smiles)
tpsa = rdMolDescriptors._CalcTPSAContribs(mol)
drawer = rdMolDraw2D.MolDraw2DCairo(300,300)
fig = SimilarityMaps.GetSimilarityMapFromWeights(mol,weights=tpsa,draw2d=drawer)
drawer.FinishDrawing()
png_data = drawer.GetDrawingText()
filename = "output.png"
path = os.path.abspath(filename)
with open(filename, "wb") as f:
f.write(png_data)
return {"image_file": f"file:///{path}"}
@mcp.tool()
def mol_tpsa(smiles):
"""
SMILESからrdkit molオブジェクトに変換をし、TPSAを計算する。
"""
mol = Chem.MolFromSmiles(smiles)
tpsa = Descriptors.TPSA(mol)
return tpsa
if __name__ == "__main__":
mcp.run(transport="stdio")
LM Studioのmcp.jsonの設定
LM Studioを起動し、”Program”を選択し”install”を押すとmcp.jsonが編集できるので、以下のように記述する。パスの記述は、上記で作成したrdkit_mcpフォルダのパスとなる。
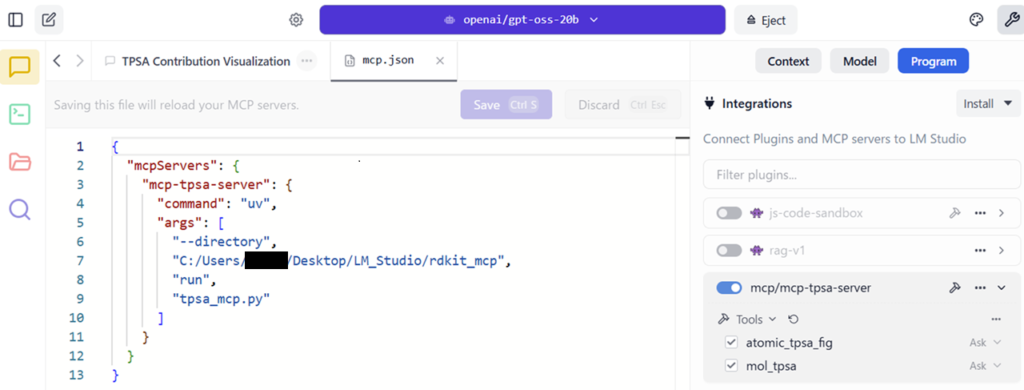
“Save”ボタンを押し、mcp/mcp-tpsa-serverをONにする。
Chatをはじめる
TPSAの計算:
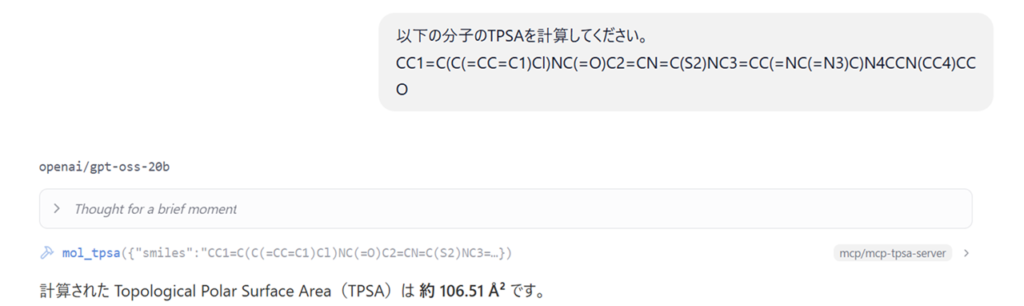
mol_tpsa関数を用いて、TPSAの計算が実行されています。
各原子のTPSAに対する寄与の計算:
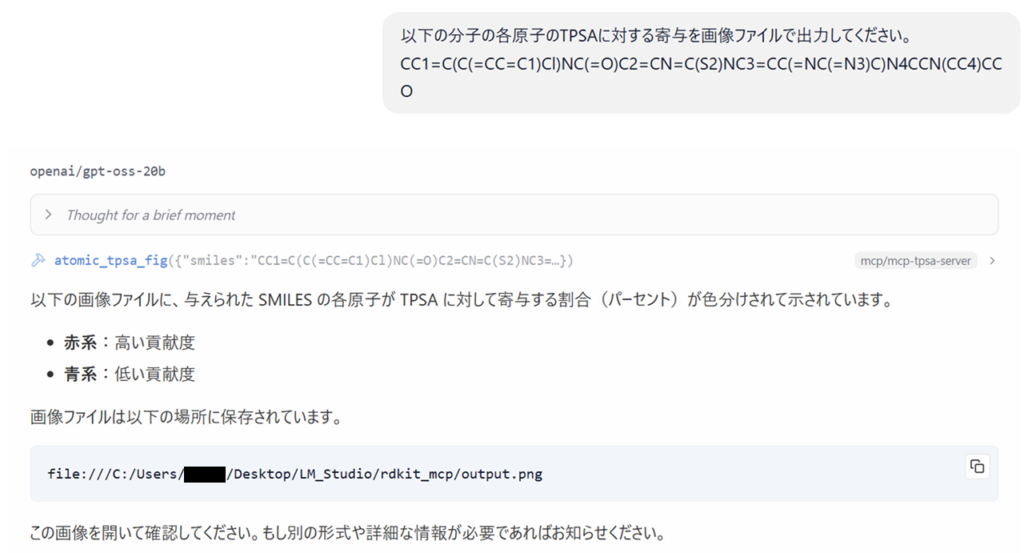
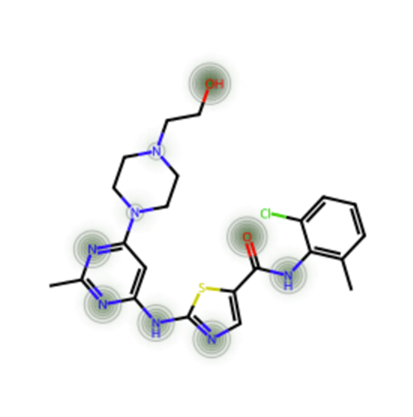
ブラウザで保存場所を開くと以下の画像が表示されます。
色分けの説明は、間違っていますね。
RDKitのスクリプトに少し手を加えるだけでMCPサーバーの構築ができます。
また、LM Studioを使えばgpt-ossとMCPサーバーの連携も簡単ですので、ローカル環境でいろいろな創薬システムの構築を楽しむことができると思います。
Category: AI創薬関連