今回は、LigandScoutを使ったdual inhibitorのスクリーニングに関する以下の文献を紹介したいと思います。また、実際に文献の一部をトレースしたいと思います。ただしLigandScoutのバージョンの違いなどがあり、全く同じようにはトレースできていませんので、その点はご了承ください。
Discovery of the first dual inhibitor of the 5-lipoxygenase-activating protein and soluble epoxide hydrolase using pharmacophore-based virtual screening.
Temml V, Garscha U, Romp E, Schubert G, Gerstmeier J, Kutil Z, Matuszczak B, Waltenberger B, Stuppner H, Werz O, Schuster D.
Sci Rep. 2017 Feb 20;7:42751. doi: 10.1038/srep42751
One-drug-hits-one-target approachは重要な戦略なのですが、近年は、
polypharmacological approachも注目されています。ここで大切なことは複数の標的タンパク質を1剤で阻害するための設計手法だといえます。
本文献では、炎症にかかわる2つのタンパク質、5-lipoxygenase-activating protein (FLAP) とsoluble epoxide hydrolase (sEH) を共に阻害する化合物の探索をLigandScoutを用いて実施しています。
著者らは、下図に示す既存の11個のFLAP阻害剤を用いてファーマコフォアの構築とテストを行っています。
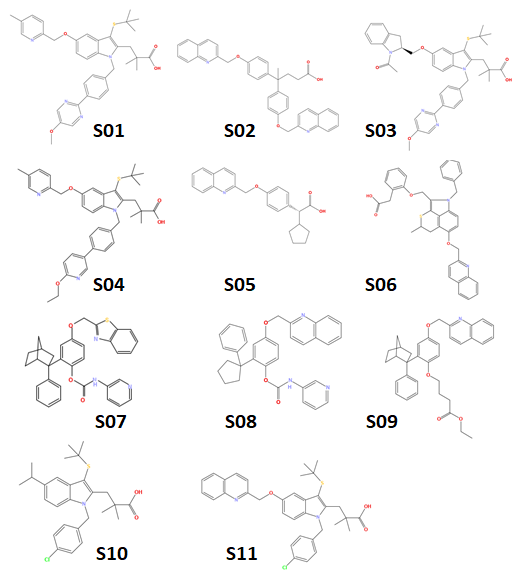
文献では、2種類のファーマコフォアモデル、Flap1,とFlap2を構築しています。Flap1はS10,S11を用いて、そしてFlap2はS07,S08,S09を用いて構築されています。
本記事では、Flap2の構築を行いたいと思います。本文献で用いられているLigandScoutのバージョンは、3.12ですが、本記事では、4.3となります。
ファーマコフォアモデルFlap2の構築:
LigandScoutを起動し、”Ligand-Based” Perspectiveに移動します。
“Ligand-Set”→”Add Molecules”→”To Test-Set”からSall.sdfを選択します。
Sall.sdfには、S01からS11の化合物が入っています。
S07,S08,S09からファーマコフォアモデルを作成したいので、この3つをTrainingと設定します。
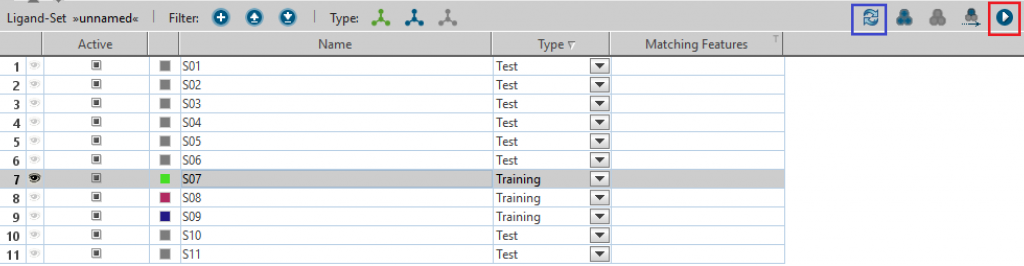
青四角を押して、配座生成を行い、赤四角を押してファーマコフォアモデルを構築します。Scoring functionは”Pharmacophore-Fit”、Pharmacophore typeは”Shared feature pharmacophore”を選択しました。
下図のようなファーマコフォアモデルが生成されました。本文献のFig.1Bを見ながらFeatureを選別しました。
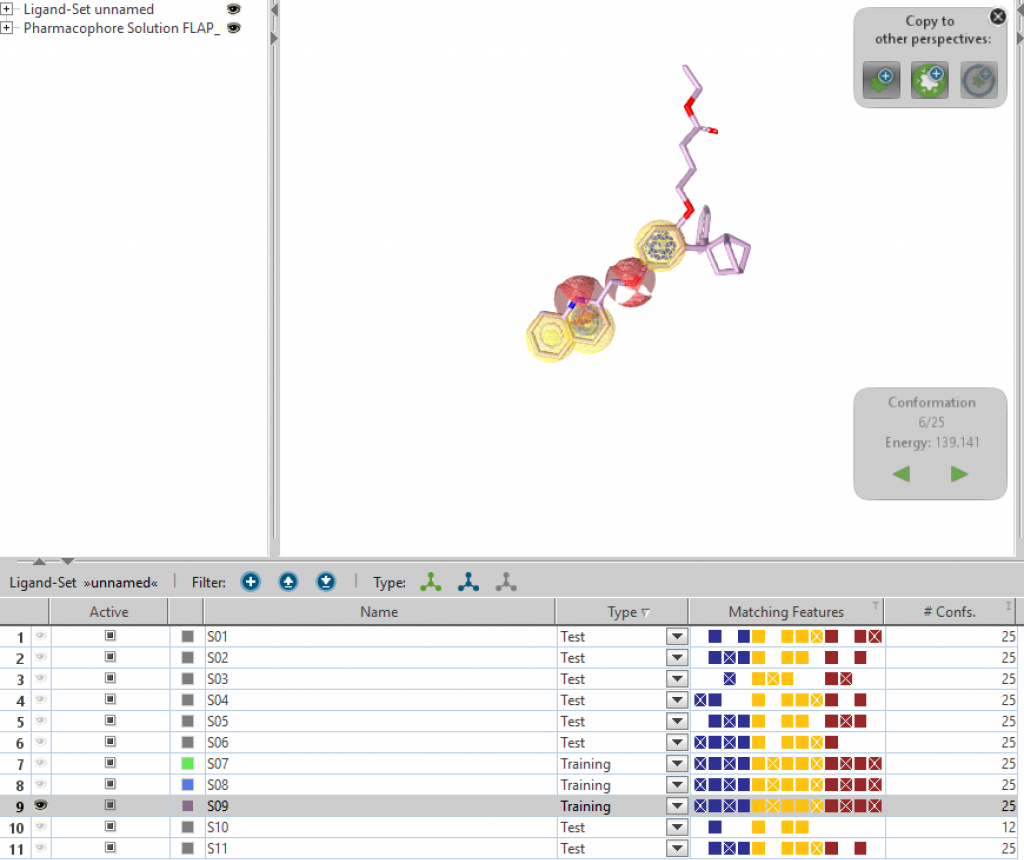
排除体積ですが、化合物S01からS11を選択した状態で”Pharmacophore”→”Add Exclusion Volumes Coat”により追加をしました。この操作は本文献とは異なるかもしれません。
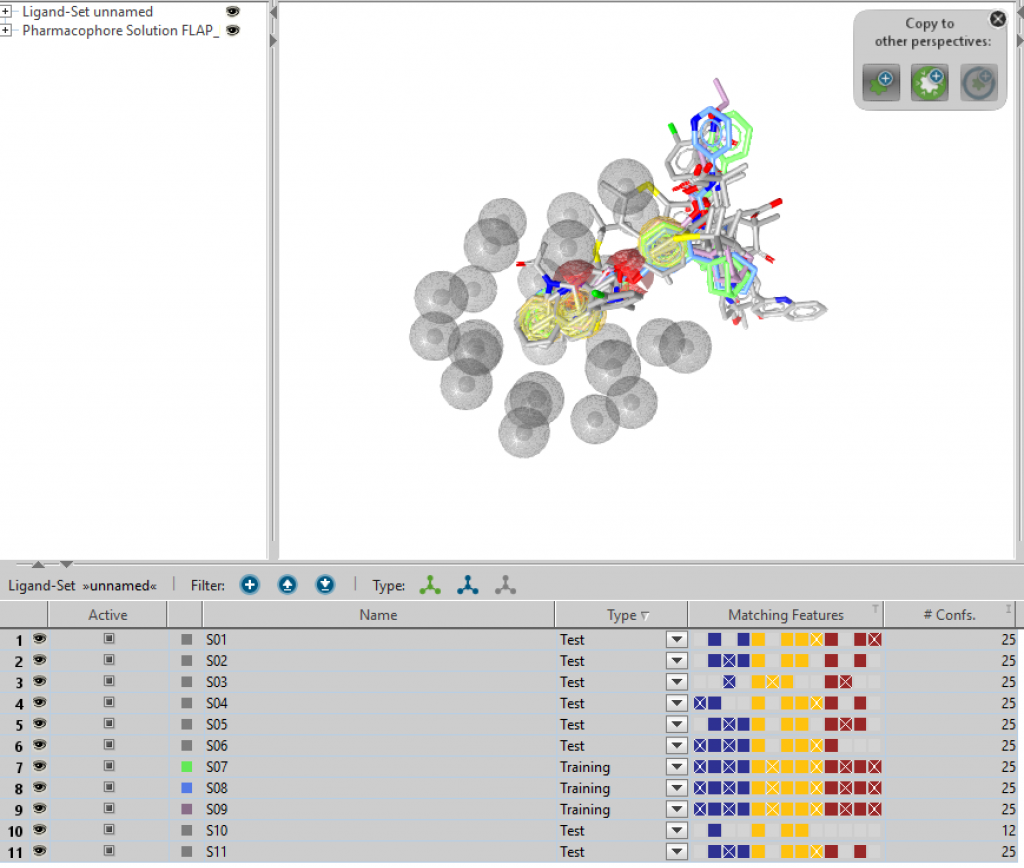
ファーマコフォアモデルFlap2の評価:
”Ligand-Based” Perspectiveで構築したFlap2を”Screening” Perspecitveにコピーします。Sall.sdfからSall.ldbの作成を行った後、Flap2を用いてファーマコフォア探索したところ、9化合物(S01,S02,S04,S05,S06,S07,S08,S09,S11)がヒットしました。本文献においてもFlap2により11化合物中9化合物がヒットしたと記載されています。
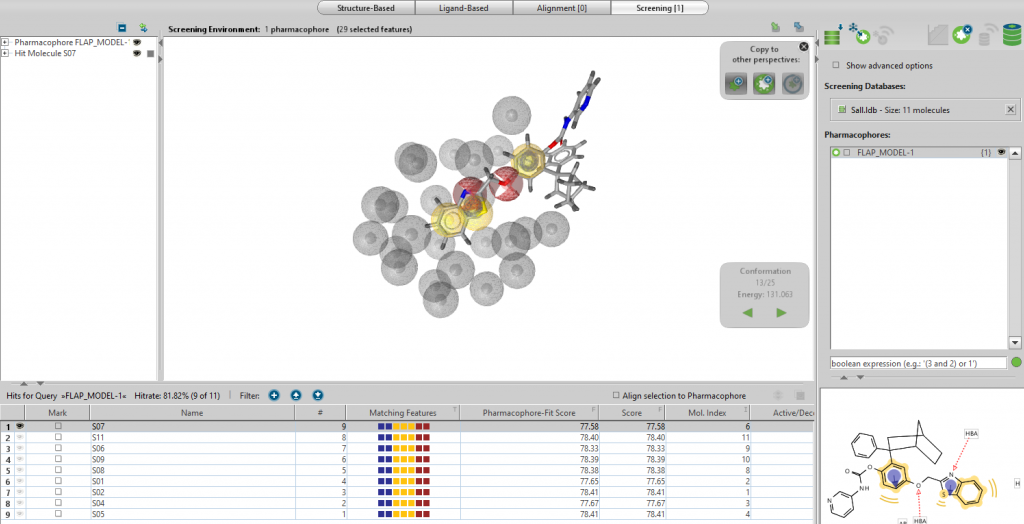
Compoud 5とFlap2のマッチング:
文献では、Flap1とFlap2を用いて、SPECS virtual library (202,920 compounds, version 2012)に対してスクリーニングを行っています。その後、Flap1でヒットした10化合物とFlap2でヒットした10化合物を実際のアッセイで評価を行ったところ、FLAPとsEHを強く阻害する化合物としてCompound 5が得られています(IC50 FLAP = 0.2uM, IC50 sEH = 0.02uM)。
ここでは、本記事で作成したFlap2が、Compound5をヒット化合物として認識するか否かをチェックしてみました。
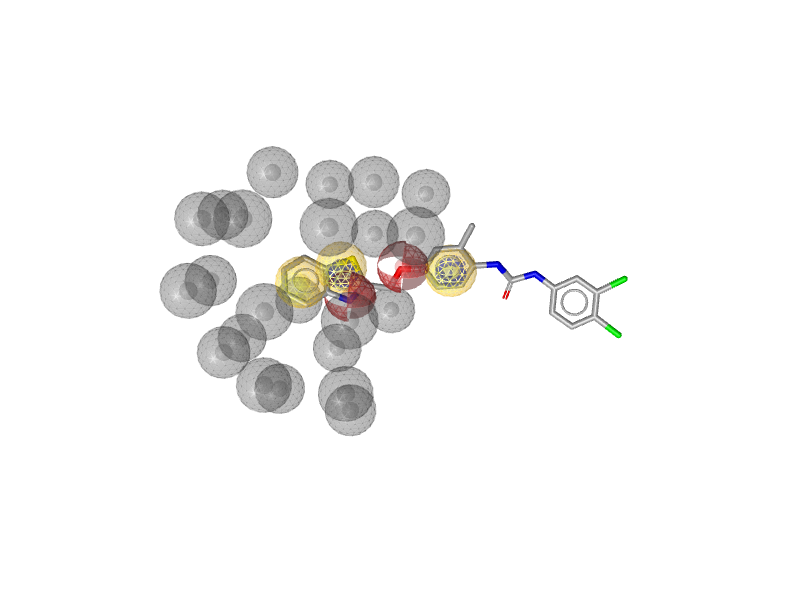
“Screening” Perspectiveで確認したのですが、Compound 5を認識することができました。
Compound 5とsEHファーマコフォアモデルとのマッチング:
本文献で用いているsEHファーマコフォアモデルは、別の文献で作成されたものが利用されています。
本記事では、本文献中のFig.4A,Bを見ながら、sEHファーマコフォアモデルを構築し、Compound 5とのマッチングを確認しました。
“Structure-Based” Perspectiveにおいて、sEHのPDB code 3antを入力します。黄色ボックスで囲まれた阻害剤をクリックし、ファーマコフォア生成を行います。以下の様なファーマコフォアが得られます。
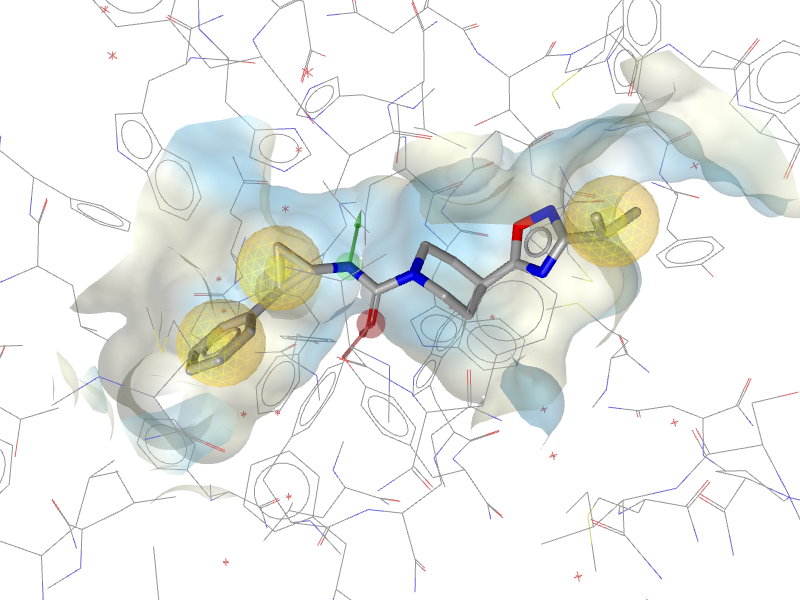
Compound 5と本sEHファーマコフォアモデルのマッチングを行いました。
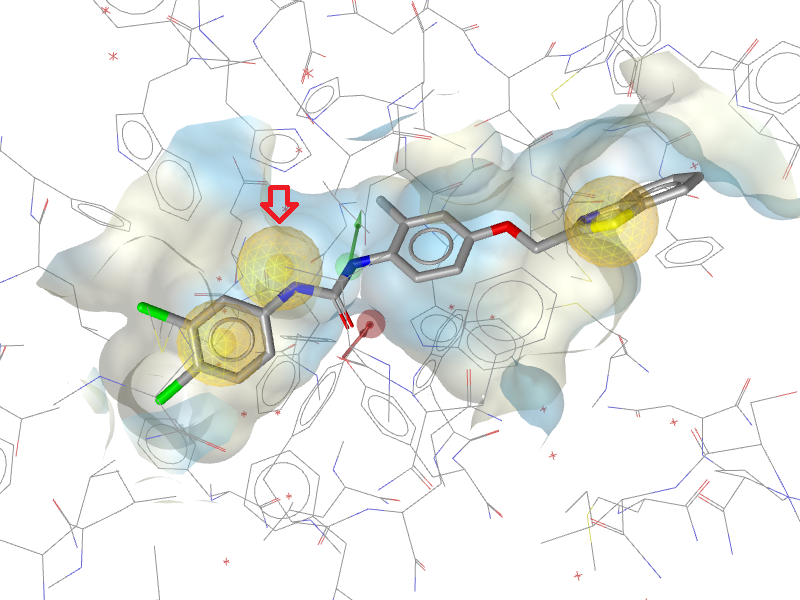
赤矢印のFeatureを無視すると文献中のFig.4Bに近い図が得られました。
ファーマコフォアを用いたdual inhibitorの探索は、polypharmacological approachを展開する上で合理的な手段の1つだと言えます。
本記事では、文献の内容を正確にトレースは出来ていませんが、ここで構築したファーマコフォアはFLAP阻害剤の探索に利用できると思われます。
次回、本記事で構築したファーマコフォアを用いて、SPECS virtual libraryに対するファーマコフォア探索を実際にやってみたいと思っています。
Category: LigandScout